スウェーデン王立科学アカデミーは、2017年のノーベル化学賞を”溶液中の生体分子の構造を高い分解能で決定するためのクライオ電子顕微鏡の開発”に貢献したとして、ジャック・デュボシェ、ヨアヒム・フランク、リチャード・ヘンダーソンの3氏に授与すると決定しました。Scientific Background on the Nobel Prize in Chemistry 2017: The Development of Cryo-Electron Microscopy
クライオEMは、Gatan社の直接検出カメラ (K3® および K2®) のテクノロジとイノベーションによって牽引されています。
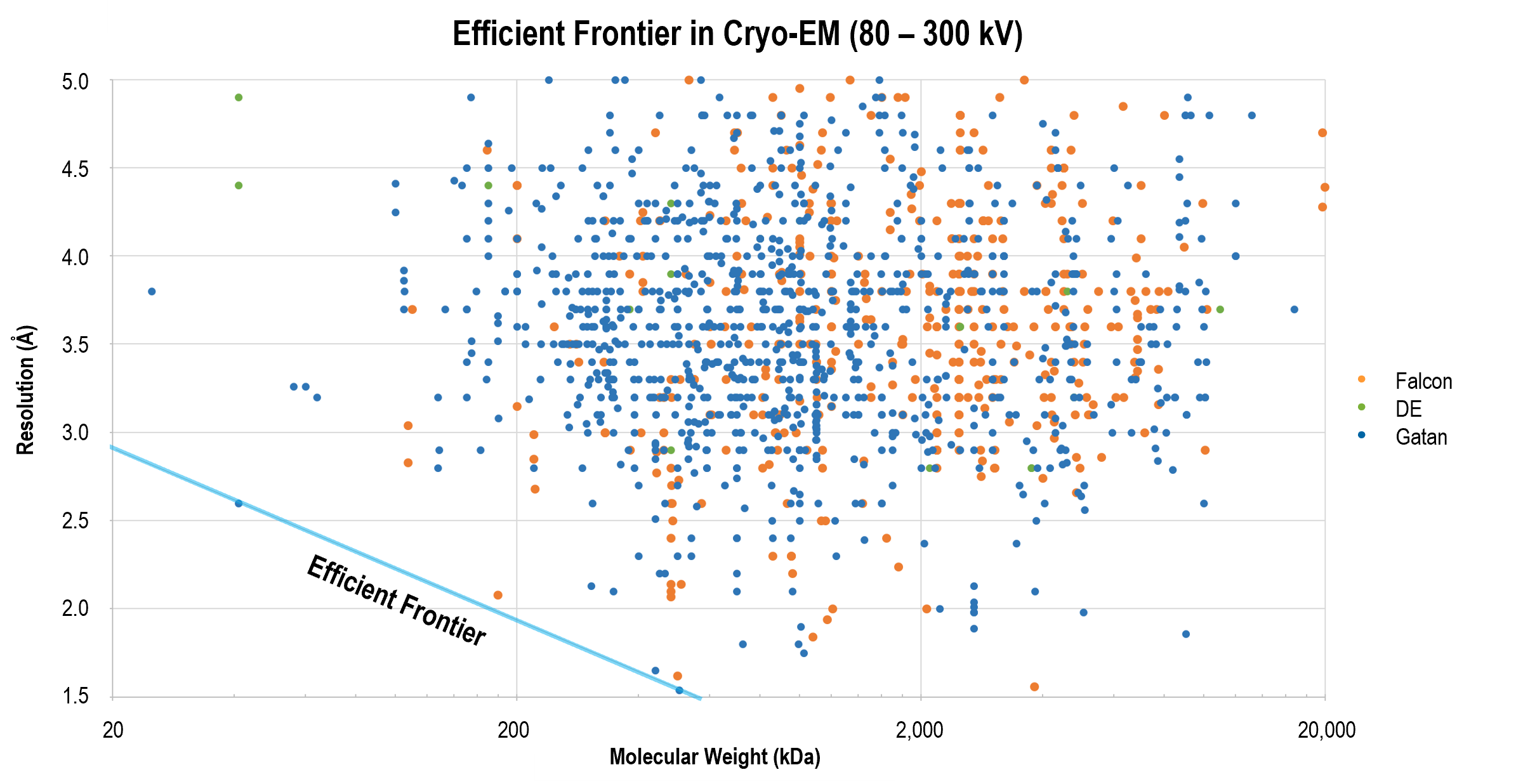
分解能の効率的フロンティア メリット クライオEMのワークフロー
クライオ電子顕微鏡における単粒子解析法とは?
 単粒子解析クライオ電子顕微鏡(クライオEM)は、構造生物学者が原子分解能で構造を解明するために使用する手法で広く採用されています。この手法はX線結晶構造解析法を補完するもので、結晶試料がなくても構造上の詳細が明らかになります。ガラス質(非晶質)の氷で凍結水和した試料を観察することにより、試料の超微細構造、緩衝液、および配位子分布が元の状態のまま保持されます。また、クライオEMは、90 kDaより大きい試料の研究が可能となる核磁気共鳴(NMR)を使用した構造解析を補完します。構造生物学者はクライオEMを頻繁に使用して、超分子集合体またはマシンにおける分子の相互作用のほか、ウイルス、小さな細胞器官、および高分子の生物学的複雑性を研究しています。
単粒子解析クライオ電子顕微鏡(クライオEM)は、構造生物学者が原子分解能で構造を解明するために使用する手法で広く採用されています。この手法はX線結晶構造解析法を補完するもので、結晶試料がなくても構造上の詳細が明らかになります。ガラス質(非晶質)の氷で凍結水和した試料を観察することにより、試料の超微細構造、緩衝液、および配位子分布が元の状態のまま保持されます。また、クライオEMは、90 kDaより大きい試料の研究が可能となる核磁気共鳴(NMR)を使用した構造解析を補完します。構造生物学者はクライオEMを頻繁に使用して、超分子集合体またはマシンにおける分子の相互作用のほか、ウイルス、小さな細胞器官、および高分子の生物学的複雑性を研究しています。
クライオEMの観察時には、同一でありながらランダムに配向した何千から何十万もの粒子(分子)の高分解能像を各試料から記録するために、透過型電子顕微鏡(TEM)が使用されます。次に、複数の3次元的な分子配向を区別するため、これらの画像は画像分類アルゴリズムを使用して分類、整列、平均化されます。クライオEMは、1.5Å以下の分解能 で分子構造を解明できます。これは、数年前には想定し得なかった分解能レベルです。
クライオEMの分解能のこの急速な向上は「分解能革命」と呼ばれ、直接検出カメラの直接的結果です。電子顕微鏡の従来のカメラは、シンチレーターを使用して電子の像を光の像に変換し、光ファイバーを使用してその画像をアナログ記録用CCDまたはCMOS画像センサーに転送します。観察結果が変換されて記録されるまでの間に高分解能像の細部が失われ、長い間クライオEMは真の潜在能力を発揮できませんでした。
従来型カメラでは変換ステップで細部の情報が失われるため、直接検出カメラでは電子の像を直接測定しています。Gatan K3 カメラは、像を記録するために 超解像技術を備えた電子カウンティング を採用した独自の直接検出型カメラです。この技術では、アナログの読み取りノイズを排除し、個々の電子をリアルタイムで認識およびカウントします。Gatan社の直接検出型カメラでは、比類ない0.5ナイキストにおける 量子検出効率 (DQE) を実現しています。
分解能の効率的フロンティア
Gatanの直接検出型カメラは、多くの場合、GIF Quantum LSイメージングフィルタと組み合わされ、分解能の効率的フロンティアを再定義する成功的な結果を出し続けています。最先端の結果を示す論文では、最新のデータ取得および画像処理方法を備えたこれらの製品が、βガラクトシダーゼの2.2オングストローム構造 など、より小さな分子をより高い分解能で再構築する最前線にいることを示しています。
グラフは単粒子クライオ電子顕微鏡法を用いた論文における分解能と分子量との間の関係を示しています。Gatanの直接検出型カメラは、様々な分子量に対して分解能の限界を拡げる多くの構造解析に用いられています。
単粒子解析クライオEMのメリット
単粒子解析クライオEMでは現在、X線結晶構造解析と同等の分解能の構造が得られるようになっており、その技術には構造生物学者の 注目を集める 数多くの独自のメリットが存在します。:
| 能力 | メリット |
|---|---|
| 自然な水和状態で構造を観察 | 標本や緩衝液の濃度など、生物学的に関連した環境に試料を維持 |
| より大きな集合体の研究が可能 | 不均一、準安定性、または結晶化が非常に困難である複数のサブユニットを含む150 kDaより大きな分子のキャラクタリゼーションに有効 |
| 原子分解能の構造を解明 | αらせん、βシートに加え、非対称側鎖、水素結合、および水分子の観察が可能 |
| 化学的な環境を制御 | さまざまな機能状態で分子を観察するために実験条件を変更可能 |
| 結晶化のステップを排除 | 時間を要する不確定な試料準備が不要、論文のパブリッシュまでの時間を短縮 |
単粒子解析クライオEMのワークフロー
|
|
ステップ1: 精製 単粒子解析クライオEMで分子を研究するには、高品質の3次元再構築を行うために試料は精製され構造的に完全である必要があります。理想的には、試料を生化学的に活性な状態に保つ緩衝液に保持する必要があります。試料内の分子は、顕微鏡で観察を行うのに十分高い濃度である必要がありますが、それらが凝固するほど高くならないようにします。最終的に、関心のある分子の均一の立体配座状態を促進するよう実験条件は最適化されます。 |
|
|
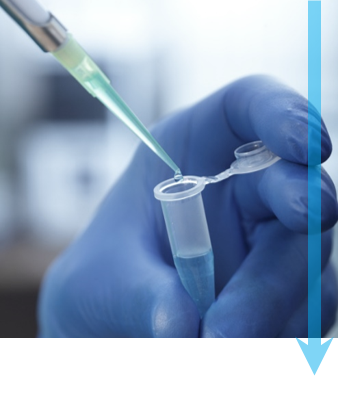
ステップ2: 急速凍結 各試料は、電子顕微鏡の真空内での凍結乾燥を防ぐために凍結されます。ほぼ瞬間的な凍結は、試料の構造を破壊する水の結晶が形成されるのを防ぎます。 まず、溶液中の少量の試料をTEMグリッドに付着させてから、ろ紙で余分な液体を取り除きます。TEMグリッドを液体エタン、または液体エタンと液体プロパンの混合液に浸して試料を素早く湿らし熱を奪うことで、非晶質またはガラス質の氷を生成します。画像は、試料を付着させ液体の冷媒に浸す直前の状態の Cryoplunge® 3 システムです。 |
|
|
ステップ3: TEMへの搬送 試料を凍結したら、液体窒素温度に試料を保持する専用のTEMホルダーに移動します。試料の汚染を防ぐため、試料をホルダーに装填する際には、クライオワークステーションが試料を保護します。また、ワークステーションからTEMに移動する際には、クライオシールドがそれをカプセル化します。この図では、 クライオトランスファーホルダー がTEMへの挿入前にワークステーションから取り外された状態を示しています。 |
|
|
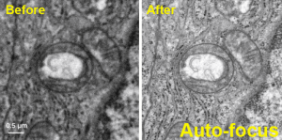
ステップ4: 試料の像観察 試料の構造は、電子線照射により損傷を受けます。通常、高分解能の構造情報が失われる前までに、合計10~30e-/Å2の電子線照射量が利用可能です。試料の損傷を防ぐには、低電子線照射量イメージング法を使用して目的の場所に移動し、像を取得する直前だけ電子線を絞ります。 電子カウンティングと超解像モード を備えたGatanの直接検出型カメラによって実現された高いDQEによって、弱い生物系試料に対しても最高のクオリティの像が取得可能です。この高いS/N比により、粒子の3次元再構築における水分子、イオン、および配位子構造を区別することができます。最高75フレーム/秒でフルフレームを保存し、後に試料の動きを補正しドリフトを最小化する、Gatanの直接検出型カメラに備えられた 分割露光 機能を利用することで像質を更に改善することが可能です。 |
|
|
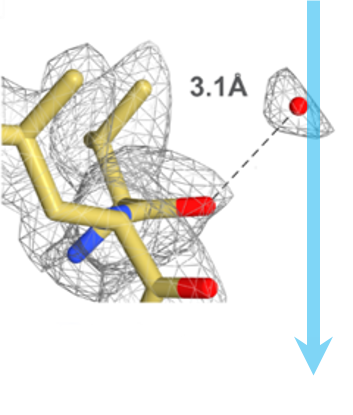
ステップ5: 解析と再構築 像の撮影が終了したら Gatan Microscopy Suite® ソフトウェアが解析をサポートしデータを様々なフォーマットへとエクスポートします。Gatan社製カメラで得られたデータは、3次元再構築と可視化のためのEMAN、Frealign、Relionといったサードパーティのソフトウェアツールにインポート可能です。左図は 2.8 Åの分解能での20Sプロテアソーム の3次元クライオEM密度マップを示しています。 |
Gatan社製カメラから得られる低電子線照射、単粒子、クライオ電子顕微鏡観察(Cryo-EM)のデータセットを効率的に、高いスループットで取得するための新たなスタンダードとなるべく開発されました。
Gatan Microscopy Suiteとしても知られるDigitalMicrographソフトウェア。デジタルカメラの制御と共に、電子線トモグラフィ、その場観察、スペクトラムイメージング、ディフラクションイメージング、その他多くのアプリケーションをサポート。
-
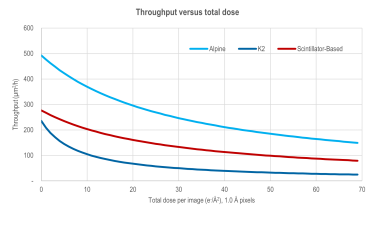 Throughput versus total dose comparison - Alpine
Throughput versus total dose comparison - Alpine
-
 Aldolase resolved to 3.07 Å at 100 keV
Aldolase resolved to 3.07 Å at 100 keV
-
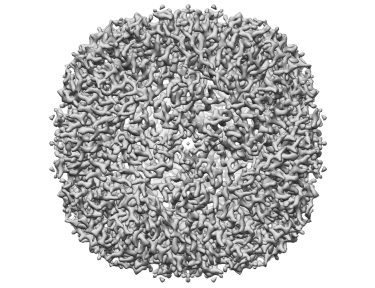 Apoferritin resolved to 2.7 Å at 100 keV
Apoferritin resolved to 2.7 Å at 100 keV
-
 Imaging molecules in their native environment: Cryo-electron tomography of PCDH15 complexes in mouse stereocilia
Imaging molecules in their native environment: Cryo-electron tomography of PCDH15 complexes in mouse stereocilia
-

 コンテクストの重要性: Cryo-ETを用いた核膜孔複合体構造に対する細胞環境の影響の解明
コンテクストの重要性: Cryo-ETを用いた核膜孔複合体構造に対する細胞環境の影響の解明
-
 Cryo EM reveals mechanisms of gating and drug modulation in 5 HT3A receptors webinar
Cryo EM reveals mechanisms of gating and drug modulation in 5 HT3A receptors webinar
-
 CryoARM / K3の組み合わせによる高分解能観察 : SerialEMとLatitude、そしてデータ取得の将来
CryoARM / K3の組み合わせによる高分解能観察 : SerialEMとLatitude、そしてデータ取得の将来
-

 構造生物学を用いたパンデミックへの備え
構造生物学を用いたパンデミックへの備え
-
 Elsa Cryo-Transfer Workstation
Elsa Cryo-Transfer Workstation
-
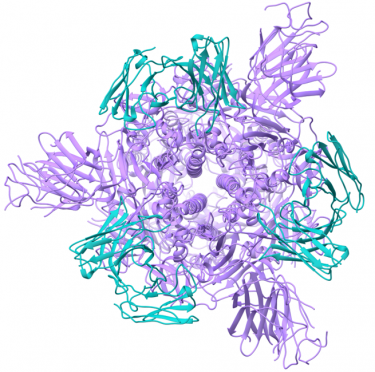 Potent neutralizing monoclonal antibodies directed to multiple epitopes on the SARS-CoV-2 spike
Potent neutralizing monoclonal antibodies directed to multiple epitopes on the SARS-CoV-2 spike
In situ structural analysis of SARS-CoV-2 spike reveals flexibility mediated by three hinges
Turoňová, B.; Sikora, M.; Schürmann, C.; Hagen, W. J. H.; Welsch, S.; Blanc, F. E. C.; von Bülow, S.; Gecht, M.; Bagola, K.; Hörner, C.; van Zandbergen, G.; Landry, J.; de Azevedo, N. T. D.; Mosalaganti, S.; Schwarz, A.; Covino, R.; Mühlebach, M. D.; Hummer, G.; Locker, J. K.; Beck, M.
Molecular architecture of the SARS-CoV-2 virus
Yao, H.; Song, Y.; Chen, Y.; Wu, N.; Xu, Sun, C.; Zhang, J.; Weng, T.; Zhang, Z.; Wu, Z.; Cheng, L,; Shi, D.; Lu, X.; Lei, J.; Crispin, M.; Shi, Y.; Li, L.; Li, S.
Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation
Wrapp, D.; Wang, N.; Corbett, K. S.; Goldsmith, J. A.; Hsieh, C. -L.; Abiona, O.; Graham, B. S.; McLellan, J. S.
Cryo-EM structure of the Ebola virus nucleoprotein–RNA complex at 3.6 Å resolution
Sugita, Y.; Matsunami, H.; Kawaoka, Y.; Noda, T.; Wolf, M.
Venugopal, H.; Mobbs, J.; Taveneau, C.; Fox, D. R.; Vuckovic, Z.; Knott, G.; Grinter, R.; Thal, D.; Mick, S.; Czarnik, C.; Ramm, G.
Chan, L. M.; Courteau, B. J.; Maker, A.; Wu, M.; Basanta, B.; Mehmood, H.; Bulkley, D.; Joyce, D.; Lee, B. C.; Mick, S.; Gulati, S.; Lander, G. C.; Verba, K. A.
Electron-counting MicroED data with the K2 and K3 direct electron detectors
Clabbers, M. T. B.; Martynawycz, M. W.; Hattne, J.; Nannenga, B. L.; Gonen, T.
Yang, T.; Xua, H.; Zoua, X.
Time-resolved cryo-EM using a combination of droplet microfluidics with on-demand jetting
Torino, S.; Dhurandhar, M.; Stroobants, A.; Claessens, R.; Efremov, R. G.
Szemán, A. J. K.; Stráner, P.; Jákli, I.; Hosogi, N.; Harmat, V.; Menyhárd, D. K.; Perczel, A.
Structural basis of actin filament assembly and aging
Oosterheert, W.; Klink, B. U.; Belyy, A.; Pospich, S.; Raunser, S.
Helical ultrastructure of the metalloprotease meprin α in complex with a small molecule inhibitor
Bayly-Jones, C.; Lupton, C. J.; Fritz, C.; Venugopal, H.; Ramsbeck, D.; Wermann, M.; Jäger,C.; Marco, A. D.; Schilling, S.; Schlenzig, D.; Whisstock, J. C.
Stern, A. M.; Yang, Y.; Meunier, A. L.; Liu, W.; Cai, Y.; Ericsson, M.; Liu, L.; Goedert, M.; Scheres, S. H. W.; Selkoe, D.J
Cryo-EM structure of the human NKCC1 transporter reveals mechanisms of ion coupling and specificity
Neumann, C.; Rosenbæk, L. L.; Flygaard, R. K.; Habeck, M.; Karlsen, J. L.; Wang, Y.; Larsen, K. L.; Gad, H. H.; Hartmann, R.; Lyons, J. A; Fenton, R. A.; Nissen, P.
Algal photosystem I dimer and high-resolution model of PSI-plastocyanin complex
Naschberger, A.; Mosebach, L.; Tobiasson, V.; Kuhlgert, S.; Scholz, M.; Perez-Boerema, A.; Ho, T. T. H.; Vidal-Meireles, A.; Takahashi, Y.; Hippler, M.; Amunts, A.
Chang, M. R.; Tomasovic, L.; Kuzmina, N. A.; Ronk, A. J.; Byrne, P. O.; Johnson, R.; Storm, N.; Olmedillas, E.; Hou, Y. J.; Schäfer, A.; Leist, S. R.; Tse, L. T.; Ke, H.; Coherd, C.; Nguyen, K.; Kamkaew, M.; Honko, A.; Zhu, Q.; Alter, G.; Saphire, E. O.; McLellan, J. S.; Griffiths, A.; Baric, R. S.; Bukreyev. A.; Marasco. W. A.
Frieg, B.; Geraets, J. A.; Strohäker, T.; Dienemann, C.; Mavroeidi, P.; Jung, B. C.; Kim, W. S.; Lee, S. J.; Xilouri, M.; Zweckstetter. M.; Schröder. G. F.
Lyu, M.; Ayala, J. C.; Chirakos, I.; Su, C. -C.; Shafer, W. M.; Yu, E. W.
Organic crystal growth: Hierarchical self-assembly involving nonclassical and classical steps
Biran, I.; Rosenne, S.; Weissman, H.; Tsarfati, Y.; Houben, L.; Rybtchinski. B.
Structure of the PAPP-ABP5 complex reveals mechanism of substrate recognition
Judge, R. A.; Sridar, J.; Tunyasunvunakool, K.; Jain, R.; Wang, J. C. K.; Ouch, C.; Xu, J.; Mafi, A.; Nile, A. H.; Remarcik, C.; Smith, C. L.; Ghosh, C.; Xu, C.; Stoll, V.; Jumper, J.; Singh, A. H.; Eaton, D.; Hao, Q.
Structures of a phycobilisome in light-harvesting and photoprotected states
Domínguez-Martín, M. A.; Sauer, P. V.; Kirst, H.; Sutter, M.; Bína, D.; Greber, B. J.; Nogales, E. ;Polívka, T.; Kerfeld, C. A.
Zhu, J.; Huang, W.; Zhao, J., Huynh, L.; Taylor, D. J.; Harris, M. E.
Miyakawa, T.; Yang, J.; Kawasaki, M.; Adachi, N.; Fujii, A.; Miyauchi, Y.; Muramatsu, T.; Moriya, T.; Senda, T.; Tanokura, M.
Kuhle, B.; Hirschi, M.; Doerfel, L. K.; Lander, G. C.; Schimmel, P.
Zhao, J.; Makhija, S.; Zhou, C.; Zhang, H.; Wang, Y.; Muralidharan, M.; Huang, B.; Cheng, Y.
Mechanistic details of CRISPR-associated transposon recruitment and integration revealed by cryo-EM
Park, J. U.; Tsai, A. W. -T.; Chen, T. H.; Peters, J. E.; Kellogg, E. H.
Specific recognition and ubiquitination of slow-moving ribosomes by human CCR4-NOT
Absmeier, E.; Chandrasekaran, V.; O'Reilly, F. J.; Stowell, J. A. W.; Rappsilber, J.; Passmore, L. A.
Cryo-electron microscopy of extracellular vesicles
Cai, K.; Sibert, B. S.; Kumar, A.; Yang, J.; Larson, M.; Thompson, K.; Wright, E. R.
Zhang, Z.; Li, Y.; Zhou, W.; Li, Y.; Chiu, W.; Cui, Y.
Peck, J. V.; Strauss, J. D.; Fay, J. F.
Cryo-EM structure of an active bacterial TIR–STING filament complex
Morehouse, B. R.; Yip, M. C. J.; Keszei, A. F. A.; McNamara-Bordewick, N. K.; Shao, S.; Kranzusch, P. J.
Maintaining the momentum in cryoEM for biological discovery
Thompson, R.; Halfon, Y.; Aspinall, L.; White, J.; Hirst, I. J.; Wang, Y.; Darrow, M.; Muench, S. P.
Orris, B.; Huynh, K. W.; Ammirati, M.; Han, S.; Bolaños, B.; Carmody, J.; Petroski, M. D.; Bosbach, B.; Shields, D. J.; Stivers, J. T.
Structural analysis of the basal state of the Artemis:DNA-PKcs complex
Watanabe, G.; Lieber, M. R.; Williams, D. R.
Flipped over U: structural basis for dsRNA cleavage by the SARS-CoV-2 endoribonuclease
Frazier, M. N.; Wilson, I. M.; Krahn, J. M.; Butay, K. J.; Dillard, L. B.; Borgnia, M. J.; Stanley, R. E.
Cryo-EM structure of the HIV-1 Pol polyprotein provides insights into virion maturation
Harrison, J. J. E. K.; Passos, D. O.; Bruhn, J. F.; Baumanlynda Tuberty, J. D.; Destefano, J. J.; Ruiz, F. X.; Lyumkis, D.; Arnold, E.
Structures and gating mechanisms of human bestrophin anion channels
Owji, A. P.; Wang, J.; Kittredge, A.; Clark, Z.; Zhang, Y.; Hendrickson, W. A.; Yang, T.
High-resolution structure determination using high-throughput electron cryo-tomography
Liu, H. -F.; Ye Zhou, Y.; Bartesaghi, A.
Structural insights into dsRNA processing by Drosophila Dicer-2–Loqs-PD
Su, S.; Wang, J.; Deng, T.; Yuan, X.; He, J.; Liu, N.; Li, X.; Huang, Y.; Wang, H. -W.; Ma, J.
Structures and mechanism of the plant PIN-FORMED auxin transporter
Ung, K. L.; Winkler, M.; Schulz, L.; Kolb, M.; Janacek, D. P.; Dedic, E.; Stokes, D. L.; Hammes, U. Z.; Pedersen, B. P.
A peroxisomal ubiquitin ligase complex forms a retrotranslocation channel
Feng, P.; Wu, X.; Erramilli, S. K.; Paulo, J. A.; Knejski, P.; Gygi, S. P.; Kossiakoff, A. A.; Rapoport, T. A.
Cryo-EM structures of SARS-CoV-2 Omicron BA.2 spike
Stalls, V.; Lindenberger, J.; Gobeil, S. M. -C.; Henderson, R.; Parks, R.; Barr, M.; Deyton, M.; Martin, M.; Janowska, K.; Huang, X.; May, A.; Speakman, M.; Beaudoin, E.; Kraft, B.; Lu, X.; Edwards, R. J.; Eaton, A.; Montefiori, D. C.; Williams, W. B.; Saunders, K. O.; Wiehe, K.; Haynes, B. F.; Acharya, P.
Structure and flexibility of the yeast NuA4 histone acetyltransferase complex
Zukin, S. A.; Marunde, M. R.; Popova, I. K.; Nogales, E.; Patel, A. B.
Structure and flexibility of the yeast NuA4 histone acetyltransferase complex
Zukin, S. A.; Marunde, M. R.; Popova, I. K.; Nogales, E.; Patel, A. B.
Martinez-Molledo, M.; Nji, E.; Reyes, N.
Role of aIF5B in archaeal translation initiation
Kazan, R.; Bourgeois, G.; Lazennec-Schurdevin, C.; Larquet, E.; Mechulam, Y.; Coureux, P. -D.; Schmitt, E.
Ion complexation waves emerge at the curved interfaces of layered minerals
Whittaker, M. L.; Ren, D.; Ophus, C.; Zhang, Y.; Waller, L.; Gilbert, B.; Banfield, J. F.
Compact IF2 allows initiator tRNA accommodation into the P site and gates the ribosome to elongation
Basu, R. S.; Sherman, M. B.; Gagnon, M. G.
Structural Basis for pH-gating of the K+ channel TWIK1 at the selectivity filter
Turney, T. S.; Li, V.; Brohawn, S. G.
Cryo-EM structures of SARS-CoV-2 Omicron BA.2 spike
Stalls, V.; Lindenberger, J.; Gobeil, S. M. -C.; Henderson, R.; Parks, R.; Barr, M.; Deyton, M.; Martin, M.; Janowska, K.; Huang, X.; May, A.;l Speakman, M.; Beaudoin, E.; Kraft, B.; Lu, X.; Edwards, R. J.; Eaton, A.; Montefiori, D. C.; Williams, W.; Saunders, K. O.; Wiehe, K.; Haynes, B. F.; Acharya, P.
Xu, Y.; Margetts, M. B.; Venugopal, H.; Menting, J. G.; Kirk, N. S.; Croll, T. I.; Delaine, C.; Forbes, B. E.; Lawrence, M. C.
Structure of S1PR2–heterotrimeric G13 signaling complex
Chen, H.; Chen, K.; Huang, W.; Staudt, L. M.; Cyster, J. G.; Li, X.
Structure of the type V-C CRISPR-Cas effector enzyme
Kurihara, N.; Nakagawa, R.; Hirano, H.; Okazaki, S.; Tomita, A.; Kobayashi, K.; Kusakizako, T.; Nishizawa, T.; Yamashita, K.; Scott, D. A.; Nishimasu, H.; Nureki, O.
Cryogenic TEM imaging of artificial light harvesting complexes outside equilibrium
Krishnaswamy, S. R.; Gabrovski, I. A.; Patmanidis, I.; Stuart, M. C. A.; de Vries A. H.; Pshenichnikov, M. S.
Assembly mechanism of the pleomorphic immature poxvirus scaffold
Hyun, J.; Matsunami, H.; Kim, T. G.; Wolf, M.
Zhao, W.; Jensen, G. J.
The giant Mimivirus 1.2 Mb genome is elegantly organized into a 30 nm helical protein shield
Villalta, A.; Schmitt, A.; Estrozi, L. F.; Quemin, E. R. J.; Alempic, J. -M.; Lartigue, A.; Pražák, V.; Belmudes, L.; Vasishtan, D.; Colmant, A. M. G.; Honoré, F. A.; Couté, Y.; Grünewald, K.; Abergel, C.
Structural and functional impact by SARS-CoV-2 Omicron spike mutations
Zhang, J.; Cai, Y.; Lavine, C. L.; Peng, H.; Zhu, H.; Anand, K.; Tong, P.; Gautam, A.; Mayer, M. L.; Rits-Volloch, S.; Wang, S.; Sliz, P.; Wesemann, D. R.; Yang, W.; Seaman, M. S.; Lu, J.; Xiao, T.; Chen, B.
Capturing the swelling of solid-electrolyte interphase in lithium metal batteries
Zhang, Z.; Li, Y.; Xu, R.; Zhou, W.; Li, Y.; Oyakhire, S. T.; Wu, Y.; Xu, J.; Wang, H.; Yu, Z.; Boyle, D. T.; Huang, W.; Ye, Y.; Chen, H.; Wan, J.; Bao, Z.; Chiu, W.; Cui, Y.
Nuclear pores dilate and constrict in cellulo
Zimmerli, C. E.; Allegretti, M.; Rantos, V.; Goetz, S. K.; Obarska-Kosinska, A.; Zagoriy, I.; Halavatyi, A.; Hummer, G.; Mahamid, J.; Kosinski, J.; Beck, M.
Scaffolding protein CcmM directs multiprotein phase separation in β-carboxysome biogenesis
Zang, K.; Wang, H.; Hartl, F. U.; Hayer-Hartl, M.
Alegre, K. O.; Paknejad, N.; Su, M.; Lou, J. -S.; Huang, J.; Jordan, K. D.; Eng, E. T.; Meyerson, J. R.; Hite, R. K.; Huang, X. -Y.
Emmanuel, S. N.; Smith, J. K.; Hsi, J.; Tseng, Y. -S.; Kaplan, M.; Mietzsch, M.; Chipman, P.; Asokan, A.; Robert McKenna, R.; Agbandje-McKenna, M.
Yu, H.; Hamaguchi, T.; Nakajima, Y.; Kato, K.; Kawakami, K.; Akita, F.; Yonekura, K.; Shen, J. -R.
Peng, Q.; Peng, R.; Yuan, B.; Zhao, J.; Wang, M.; Wang, X.; Wang, Q.; Sun, Y.; Fan, Z.; Qi, J.; Gao, G. F.; Shi, Y.
A simple pressure-assisted method for MicroED specimen preparation
Zhao, J.; Xu, H.; Lebrette, H.; Carroni, M.; Taberman, H.; Hogbom, M.; Zou, X.
Structure-guided multivalent nanobodies block SARS-CoV-2 infection and suppress mutational escape
Koenig, P. -D.; Das, H.; Liu H.; Kümmerer, B. M.; Gohr, F. N.; Jenster, L. -M.; Schiffelers, L. D. J.; Tesfamariam, Y. M.; Uchima, M.; Wuerth, J. D.; Gatterdam, K.; Ruetalo, N.; Christensen, M. H.; Fandrey, C. I.; Normann, S.; Tödtmann, J.; M. P.; Pritzl, S.; Hanke, L.; Boos, J.; Yuan, M.; Zhu, X.; Schmid-Burgk, J. L.; Kato, H.; Schindler, M.; Wilson, I. A.; Geyer, M.; Ludwig, K. U.; Hällberg, M.; Wu, N. C.; Schmidt, F. I.
Ultrapotent antibodies against diverse and highly transmissible SARS-CoV-2 variants
Wang, L.; Zhou, T.; Zhang, Y.; Yang, E. S.; Schramm, C. A.
Cryo-EM structures of human coagulation factors V and Va
Ruben, E. A.; Rau, M. J.; Fitzpatrick, J. A. J.; Di Cera, E.
Wei, H.; Qian, X.; Xie, F.; Cui, D.
Native-like SARS-CoV-2 spike glycoprotein expressed by ChAdOx1 nCoV-19/AZD1222 vaccine
Watanabe, Y.; Mendonça, L.; Allen, E. R.; Howe, A.; Lee, M.; Allen, J. D.; Chawla, H.; Pulido, D.; Donnellan, F.; Davies, H.; Ulaszewska, M.; Belij-Rammerstorfer, S.; Morris, S.; Krebs, A. -S.; Dejnirattisai, W.; Mongkolsapaya, J.; Supasa, P.; Screaton, G. R.; Green, C. M.; Lambe, T.; Zhang, P.; Gilbert, S. C.; Crispin, M.
Simplified approach for preparing graphene oxide TEM grids for stained and vitrified biomolecules
Kumar, A.; Sengupta, N.; Dutta, S.
Shafiq, A.; Suwakulsiri, W.; Rai, A.; Chen, M.; Greening, D. W.; Zhu, H. -J.; Xu, R.; Simpson, R. J.
Structure of a microtubule-bound axonemal dynein
Walton, T.; Wu, H.; Brown, A.
Mechanism of SARS-CoV-2 polymerase stalling by remdesivir
Kokic, G.; Hillen, H. S.; Tegunov, D.; Dienemann, C.; Seitz, F.; Schmitzova, J.; Farnung, L.; Siewert, A.; Höbartner, C.; Cramer, P.
Stabilizing the closed SARS-CoV-2 spike trimer
Juraszek, J.; Rutten, L.; Blokland, S.; Bouchier, P.; Voorzaat, R.; Ritschel, T.; Bakkers, M. J. G.; Renault , L. L. R.; Langedijk, J. P. M.
Development and structural basis of a two-MAb cocktail for treating SARS-CoV-2 infections
Zhang, C.; Wang, Y.; Zhu, Y.; Liu, C.; Gu, C.; Xu, S.; Wang, Y.; Zhou, Y.; Wang, Y.; Han, W.; Hong, X.; Yang, Y.; Zhang, X.; Wang, T.; Xu, C.; Hong, Q.; Wang, S.; Zhao, Q.; Qiao, W.; Zang, J.; Kong, L.; Wang, F.; Wang, H.; Qu, D.; Lavillette, D.; Tang, H.; Deng, Q.; Xie, Y.; Cong, Y.; Huang, Z.
Yan, L.; Ge, J.; Zheng, L.; Zhang, Y.; Gao, Y.; Wang, T.; Huang, Y.; Yang, Y.; Gao, S.; Li, M.; Liu, Z.; Wang, H.; Li, Y.; Chen, Y.; Guddat, L. W.; Wang, Q.; Rao, Z.; Lou, Z.
Xu, C.; Wang, Y.; Liu, C.; Zhang, C.; Han, W.; Hong, X.; Wang, Y.; Hong, Q.; Wang, S.; Zhao, Q.; Wang, Y.; Yang, Y.; Chen, K.; Zheng, W.; Kong, L.; Wang, F.; Zuo, Q.; Huang, Z.; Cong, Y.
They spent 12 years solving a puzzle. It yielded the first COVID-19 vaccines.
Kramer, J.
Mihelc. E. M.; Baker, S. C.; Lanman, J. K.
Yuan, S.; Peng, L.; Park, J. J.; Hu, Y.; Devarkar, S. C.; Dong, M. B.; Shen, Q.; Wu, S.; Chen, S.; Lomakin, I. B.; Xiong, Y.
Zhou, T.; Tsybovsky, Y.; Gorman, J.; Rapp, M.; Cerutti, G.; Chuang, G. -Y.; Katsamba, P. S.; Sampson, J. M.; Schön, A.; Bimela, J.; Boyington, J. C.; Nazzari, A.; Olia, A. S.; Shi, W.; Sastry, M.; Stephens, T.; Stuckey, J.; Teng, I. -T.; Kwong, P. D
De novo design of potent and resilient hACE2 decoys to neutralize SARS-CoV-2
Linsky, T. W.; Vergara, R.; Codina, N.; Nelson, J. W.; Walker, M. J.; Su, W.; Barnes, C. O.; Hsiang, T. -Y.; Esser-Nobis, K.; Yu, K.; Reneer, B.; Hou, Y. J.; Priya, T.; Mitsumoto, M.; Pong, A.; Lau, Y.; Mason, M. L.; Chen, J.; Chen, A.; Berrocal, T.; Peng, H.; Clairmont, N. S.; Castellanos, J.; Lin, Y. -R..; Josephson-Day, A.; Baric, R. S.; Fuller, D. H.; Walkey, C. D.; Ross, T. M.; Swanson, R.; Bjorkman, P. J.; Gale Jr., M.; Blancas-Mejia, L. M.; Yen, H. -L.; Silva, D. -A.
Yao, H.; Sun, Y.; Deng, Y. -Q.; Wang, N.; Tan, Y.; Zhang, N. -N.; Li, X. -F.; Kong, C.; Xu, Y. -P.; Chen, Q.; Cao, T. -S.; Zhao, H.; Yan, X.; Cao, L.; Lv, Z.; Zhu, D.; Feng, R.; Wu, N.; Zhang, W.; Hu, Y.; Chen, K.; Zhang, R. -R.; Lv, Q.; Sun, S.; Zhou, Y.; Yan, R.; Yang, G.; Sun, X.; Liu, C.; Lu, X.; Cheng, L.; Qiu, H.; Huang, X. -Y.; Weng, T.; Shi, D.; Jiang, W.; Shao, J.; Wang, L.; Zhang, J.; Jiang, T.; Lang, G.; Qin, C. -F.; Li, L.; Wang, X.
Structure-based development of human antibody cocktails against SARS-CoV-2
Wang, N.; Sun, Y.; Feng, R.; Wang, Y.; Guo, Y.; Zhang, L.; Deng, Y. -Q.; Wang, L.; Cui, Z.; Cao, L.; Zhang, Y. -J.; Li, W.; Zhu, F. -C.; Qin, C. -F.; Wang, X.
Structural analysis of full-length SARS-CoV-2 spike protein from an advanced vaccine candidate
Bangaru, S.; Ozorowski, G.; Turner, H. L.; Antanasijevic, A.; Huang, D.; Wang, X.; Torres, J. L.; Diedrich, J. K.; Tian, J. -H.; Portnoff, A. D.; Patel, N.; Massare, M. J.; Yates III, J. R.; Nemazee, D.; Paulson, J. C.; Glenn, G.; Smith, G.; Ward, A. B.
Ultrapotent human antibodies protect against SARS-CoV-2 challenge via multiple mechanisms
Tortorici, M. A.; Beltramello, M.; Lempp, F. A.; Pinto, D.; Dang, H. V.; Rosen, L. E.; McCallum, M.; Bowen, J.; Minola, A.; Jaconi, S.; Zatta, F.; De Marco, A.; Guarino, B.; Bianchi, S.; Lauron, E. J.; Tucker, H.; Zhou, J.; Peter, A.; Havenar-Daughton, C.; Wojcechowskyj, J. A.; Case, J. B.; Chen, R. E.; Kaiser, H.; Montiel-Ruiz, M.; Meury, M.; Czudnochowski, N.; Spreafico, R.; Dillen, J.; Ng, C.; Sprugasci, N.; Culap, K.; Benigni, F.; Abdelnabi, R.; Foo, S. -Y. C.; Schmid, M. A.; Cameroni, E.; Riva, A.; Gabrieli, A.; Galli, M.; Pizzuto. M. S.; Neyts, J.; Diamond, M. S.; Virgin, H. W.; Snell, G.; Corti, D.; Fink, K.; Veesler, D.;
SARS-CoV-2 structure and replication characterized by in situ cryo-electron tomography
Klein, S.; Cortese, M.; Winter, S. L.; Wachsmuth-Melm, M.; Neufeldt, C. J.; Cerikan, B.; Stanifer, M. L.; Boulant, S.; Bartenschlager, R.; Chlanda , P.
Architecture of a SARS-CoV-2 mini replication and transcription complex
Yan, L.; Zhang, Y.; Ge, J.; Zheng, L.; Gao, Y.; Wang, T.; Jia, Z.; Wang, H.; Huang, Y.; Li, M.; Wang, Q.; Ra, Z.; Lou, Z.
Du, S.; Cao, Y.; Zhu, Q.; Yu, P.; Qi, F.; Wang, G.; Du, X.; Bao, L.; Deng, W.; Zhu, H.; Liu, J.; Nie, J.; Zheng, Y.; Liang, H.; Liu, R.; Gong, S.; Xu, H.; Yisimayi, A.; Qin, C.
Piccoli, L.; Park, Y. -J.; Tortorici, M. A.; Czudnochowski, N.; Walls, A. C.; Beltramello, M.; Silacci-Fregni, C.; Pinto, D.; Rosen, L. E.; Bowen, J. E.; Acton, O. J.; Jaconi, S.; Guarino, B.; Minola, A.; Zatta, F.; Sprugasci, N.; Bassi, J.; Peter, A.; De Marco, A.; Nix, J. C.; Mele, F.; Jovic, S.; Rodriguez, B. F.; Gupta, S. V.; Jin, F.; Piumatti, G.; Presti, G. L.; Pellanda, A. F.; Biggiogero, M.; Tarkowski, M.; Pizzuto, M. S.; Cameroni, E.; Havenar-Daughton, C.; Smithey, M.; Hong, D.; Lepori, V.; Albanese, E.; Ceschi, A.; Bernasconi, E.; Elzi, L.; Ferrari, P.; Garzoni, C.; Riva, A.; Snell, G.; Sallusto, F.; Fink, K.; Virgin, H. W.; Lanzavecchia, A.; Corti, D.; Veesler, D.
Guo, L.; Bi, W.; Wang, X.; Xu, W.; Yan, R.; Zhang, Y.; Zhao, K.; Li, Y.; Zhang, M.; Cai, X.; Jiang, S.; Xie, Y.; Zhou, Q.; Lu, L.; Dang, B.
Free fatty acid binding pocket in the locked structure of SARS-CoV-2 spike protein
Toelzer, C.; Gupta, K.; Yadav, S. K. N.: Borucu, U.; Davidson, A. D.; Williamson, M. K.; Shoemark, D. K.; Garzoni, F.; Staufer, O.; Milligan, R.; Capin, J.; Mulholland, A. J.; Spatz, J.; Fitzgerald, D.; Berger, I.; Schaffitzel, C.
An ultrapotent synthetic nanobody neutralizes SARS-CoV-2 by stabilizing inactive spike
Schoof, M.; Faust, B.; Saunders, R. A.; Sangwan, S.; Rezelj, V.; Hoppe, N.; Boone, M.; Billesbølle, C. B.; Puchades, C.; Azumaya, C. M.; Kratochvil, H. T.; Zimanyi, M.; Deshpande, I.; Liang, J.; Dickinson, S.; Nguyen, H. C.; Chio, C. M.; Merz, G. E.; Thompson, M. C.; Diwanji, D.; Schaefer, K.; Anand, A. A.; Dobzinski, N.; Zha, B. S.; Simoneau, C. R.; Leon, K.; White, K. M.; Chio, U. S.; Gupta, M.; Jin, M.; Li, F.; Liu, Y.; Zhang, K.; Bulkley, D.; Sun, M.; Smith, A. M.; Rizo, A. N.; Moss, F.; Brilot, A. F.; Pourmal, S.; Trenker, R.; Pospiech, T.; Gupta, S.; Barsi-Rhyne, B.; Belyy, V.; Barile-Hill, A. W.; Nock, S.; Liu, Y.; Krogan, N. J.; Ralston, C. Y.; Swaney, D. L.; García-Sastre, A.; Ott, M.; Vignuzzi, M.; QCRG Structural Biology Consortium; Walter, P.; Manglik, A.
Custódio, T. F.; Das, H.; Sheward, D. J.; Hanke, L.; Pazicky, S.; Pieprzyk, J.; Sorgenfrei, M.; Schroer, M. A.; Gruzinov, A. Y.; Jeffries, C. M.; Graewert, M. A.; Svergun, D. I.; Dobrev, N.; Remans, K.; Seeger, M. A.; McInerney, G. M.; Murrell, B.; Hällberg, B. M.; Löw, C.
The architecture of inactivated SARS-CoV-2 with postfusion spikes revealed by cryo-EM and cryo-ET
Liu, C.; Mendonça, L.; Yang, Y.; Gao, Y.; Shen, C.; Liu, J.; Ni, T.; Ju, B.; Liu, C.; Tang, X.; Wei, J.; Ma, X.; Zhu, Y.; Liu, W.; Xu, S.; Liu, Y.; Yuan, J.; Wu, J.; Liu, Z.; Zhang, Z.; Liu, L.; Wang, P.; Zhang, P.
Zhou, T.; Teng, I. -T.; Olia, A. S.; Cerutti, G.; Gorman, J.; Nazzari, A.; Shi, W.; Tsybovsky, Y.; Wang, L.; Wang, S.; Zhang, B.; Zhang, Y.; Katsamba, P. S.; Petrova, Y.; Banach, B. B.; Fahad, A. S.; Liu, L.; Lopez Acevedo, S. N.; Madan, B.; de Souza, M. O.; Pan, X.; Wang, P.; Wolfe, J. R.; Yin, M.; Ho, D. D.; Phung, E.; DiPiazza, A.; Chang, L. A.; Abiona, O. M.; Corbett, K. S.; DeKosky, B. J.; Graham, B. S.; Mascola, J. R.; Misasi, J.; Ruckwardt, T.; Sullivan, N. J.; Shapiro, L.; Kwong, P. D.
De novo design of picomolar SARS-CoV-2 miniprotein inhibitors
Cao, L.; Goreshnik, I.; Coventry, B.; Case, J. B.; Miller, L.; Kozodoy, L.; Chen, R. R.; Carter, L.; Walls, A. C.; Park, Y. -J.; Strauch, E. -M.; Stewart, L.; Diamond, M. S.; Veesler, D.; Baker, D.
SARS-CoV-2 neutralizing antibody structures inform therapeutic strategies
Barnes, C. O.; Jette, C. A.; Abernathy, M. E.; Dam, K. -M., A.; Esswein, S. R.; Gristick, H. B.; Malyutin, A. G.; Sharaf, N. G.; Huey-Tubman, K. E.; Lee, Y. E.; Robbiani, D. F.; Nussenzweig, M. C.; West Jr., A. P.; Bjorkman, P. J.
In situ structural analysis of SARS-CoV-2 spike reveals flexibility mediated by three hinges
Turonova, B.; Sikora, M.; Schurmann, C.; Hagen, W. J. H.; Welsch, S.; Blanc, F. E. C.; von Bulow, S.; Gecht, M.; Bagola, K.; Horner, C.; van Zandbergen, G.; Landry, J.; de Azevedo, N. T. D.; Mosalaganti, S.; Schwarz, A.; Covino, R.; Muhlebach, M. D.; Hummer, G.; Locker, J, K,; Beck, M.
InsteaDMatic: Towards cross-platform automated continuous rotation electron diffraction
Roslova, M.; Smeets, S.; Wang, B.; Thersleff, T.; Xu, H.; Zou, X.
Broad host range of SARS-CoV-2 and the molecular basis for SARS-CoV-2 binding to cat ACE2
Wu, L.; Chen, Q.; Liu, K.; Wang, J.; Han, P.; Zhang, Y.; Hu, Y.; Meng, Y.; Pan, X.; Qiao, C.; Tian, S.; Du, P.; Song, H.; Shi, W.; Qi, J.; Wang, H. -W.; Yan, J.; Gao, G. F.; Wang, Q.
Structure-based design of prefusion-stabilized SARS-CoV-2 spikes
Hseih, C. -L.; Goldsmith, J. A.; Schaub, J. M.; Divenere, A. M.; Kuo, H. -C.; Javanmardi, K.; Le, K. C.; Wrapp, D.; Lee, A. G.; Liu, Y.; Chou, C. -W.; Byrne, P. O.; Hjorth, C. K.; Johnson, N. V.; Ludes-Meyers, J.; Nguyen, A. W.; Park, J.; Wang, N.; Amengor, D.; Lavinder, J. J.; Ippolito, G. C.; Maynard, J. A.; Finkelstein, I. J.; McLellan, J. S.
Structural basis for neutralization of SARS-CoV-2 and SARS-CoV by a potent therapeutic antibody
Lv, Z.; Deng, Y. -Q.; Ye, Q.; Cao, L.; Sun, C. -Y.; Fan, C.; Huang, W.; Sun, S.; Sun, Y.; Zhu, L.; Chen, Q.; Wang, N.; Nie, J.; Cui, Z.; Zhu, D.; Shaw, N.; Li, X. -F.; Li, Q.; Xie, L.; Wang, Y.; Rao, Z.; Qin, C. -F.; Wang, X.
Chen, J.; Malone, B.; Llewellyn, E.; Grasso, M.; Shelton, P. M. M.; Olinares, P. D. B.; Maruthi, K.; Eng, E. T.; Vatandaslar, H.; Chait, B. T.; Kapoor, T. M.; Darst, S. A.; Campbell, E. A.
Receptor binding and priming of the spike protein of SARS-CoV-2 for membrane fusion
Benton, D. J.; Wrobel, A. G.; Xu, P.; Roustan, C.; Martin, S. R.; Rosenthal, P. B.; Skehel, J. J.; Gamblin, S. J.
SARS-CoV-2 Nsp1 binds the ribosomal mRMA channel to inhibit translation
Schubert, K.; Karousis, E. D.; Jomaa, A.; Scaiola, A.; Echeverria, B.; Gurzeler, L. -A.; Leibundgut, M.; Thiel, V.; Mühlemann, O.; Ban, N.
Neutralization of SARS-CoV-2 by destruction of the prefusion spike
Huo, J.; Zhao, Y.; Ren, J.; Zhou, D.; Duyvesteyn, H. M. E.; Ginn, H. M.; Carrique, L.; Malinauskas, T.; Ruza, R. R.; Shah, P. N. M.; Tan, T. K.; Rijal, P.; Coombes, N.; Bewley, K. R.; Tree, J. A.; Radecke, J.; Paterson, N. G.; Supasa, P.; Mongkolsapaya, J.; Screaton, G. R.; Carroll, M.; Townsend, A.; Fry, E. E.; Owens, R. J.; Stuart, D. I.
An alpaca nanobody neutralizes SARS-CoV-2 by blocking receptor interaction
Hanke, L.; Perez, L. V.; Sheward, D. J.; Das, H.; Schulte, T.; Moliner-Morro, A.; Corcoran, M.; Achour, A.; Hedestam, G. B.; Hällberg, B. M.; Murrell, B.; McInerney, G. M.
Structural basis for translational shutdown and immune evasion by the Nsp1 protein of SARS-CoV-2
Thoms, M.; Buschauer, R.; Ameismeier, M.; Loepke, L.; Denk, T.; Hirschenberger, M.; Kratzat, H.; Hayn, M.; Mackens-Kiani, T.; Cheng, J.; Straub, J. H.; Sturzel, C. M.; Frohlich, T.; Berninghausen, O.; Becker, T.; Kirchhoff, F.; Sparrer, K. M.; Beckmann, R.
Molecular architecture of the SARS-CoV-2 virus
Yao, H.; Song, Y.; Chen, Y.; Wu, N.; Xu, J.; Sun, C.; Zhang, J.; Weng, T.; Zhang, Z.; Wu, Z.; Cheng, L.; Shi, D.; Lu, X.; Lei, J.; Crispin, M.; Shi, Y.; Li, L.; Li, S.
Studies in humanized mice and convalescent humans yield a SARS-CoV-2 antibody cocktail
Hansen, J.; Baum, A.; Pascal, K. E.; Russo, V.; Giordano, S.; Wloga, E.; Fulton, B. O.; Yan, Y.; Koon, K.; Patel, K.; Chung, K. M.; ermann, A.; Ullman, E.; Cruz, J.; Rafique, A.; Huang, T.; Fairhurst, J.; Libertiny, C.; Malbec, M.; Lee, W. -Y.; Welsh, R.; Farr, G.; Pennington, S.; Deshpande, D.; Cheng, J.; Watty, A.; Bouffard, P.; Babb, R.; Levenkova, N.; Chen, C.; Zhang, B.; Hernandez, A. R.; Saotome, K.; Zhou, Y.; Franklin, M.; Sivapalasingam, S.; Lye, D. C.; Weston, S.; Lopgue, J.; Haupt, R.; Frieman, M.; Chen., G.; Olson, W.; Murphy, A. J.; Stahl, N.; Yancopoulos, G. D.; Kyratsous, C. A.
Barnes, C. O.; West Jr., A. P.; Huey-Tubman, K. E.; Hoffmann, M. A. G.; Sharaf, N. G.; Hoffman, P. R.; Koranda, N.; Gristick, H. B.; Gaebler, C.; Muecksch, F.; Lorenzi, J. C. C.; Finkin, S.; Hägglöf, T.; Hurley, A.; Millard, K. G.; Weisblum, Y.; Schmidt, F.; Hatziioannou, T.; Bieniasz, P. D.; Caskey, M.; Robbiani, D. F.; Nussenzweig, M. C.; Bjorkman. P. J.
A neutralizing human antibody binds to the N-terminal domain of the spike protein of SARS-CoV-2
Chi, X.; Yan, R.; Zhang, G.; Zhang, Z.; Hao, M.; Zhang, Z.; Fan, P.; Dong, X.; Yang, Y.; Chen, Z.; Guo, Y.; Zhang, J.; Li, Y.; Song, X.; Cheng, Y.; Xia, L.; Fu, L.; Hou, L.; Xu, J.; Yu, X.; Li, J.; Zhou, Q.; Chen, W.
Structural basis for the neutralization of SARS-CoV-2 by an antibody from a convalescent patient
Zhou, D.; Duyvesteyn, H. M. E.; Chen, C. -P.; Huang, C. -G.; Chen, T. -H.; Shih, S. -R.; Lin, Y. -C.; Cheng, C. -Y.; Cheng, S. -H.; Huang, Y. -C.; Lin, T. -Y.; Ma, C.; Huo, J.; Carrique, L.; Malinauskas, T.; Ruza, R. R.; Shah, P. N. M.; Tan, T. K.; Rijal, P.; Donat, R. F.; Godwin, K.; Buttigieg, K. R.; Tree, J. A.; Radecke, J.; Paterson, N. G.; Supasa, P.; Mongkolsapaya, J.; Screaton, G. R.; Carroll, M. W.; Gilbert-Jaramillo, J.; Knight, M. L.; James, W.; Owens, R. J.; Naismith, J. H.; Townsend, A. R.; Fry, E. E.; Zhao, Y.; Ren, J.; Stuart, D. I.; Huang, K. -Y. A.
A thermostable, closed SARS-CoV-2 spike protein trimer
Xiong, X.; Qu, K.; Ciazynska, K. A.; Hosmillo, M.; Carter, A. P.; Ebrahimi, S.; Ke, Z.; Scheres, S. H. W.; Bergamaschi, L.; Grice, G. L.; Zhang, Y.; The CITIID-NIHR COVID-19 BioResource Collaboration; Nathan, J. A.; Baker, S.; James, L. C.; Baxendale, H. E.; Goodfellow, I.; Doffinger, R.; Briggs, J. A. G.
Structural basis for RNA replication by the SARS-CoV-2 polymerase
Wang, Q.; Wu2, J.; Wang, H.; Gao, Y.; Liu, Q.; Mu, A.; Ji, W.; Yan, L.; Zhu, Y.; Zhu, C.; Fang, X.; Yang, X.; Huang, Y.; Gao, H.; Liu, F.; Ge, J.; Sun, Q.; Yang, X.; Xu, W.; Liu, Z.; Yang, H.; Lou, Z.; Jiang, B.; Guddat, L. W.; Gong, P.; Rao, Z.
Controlling the SARS-CoV-2 spike glycoprotein conformation
Henderson, R.; Edwards, R. J.; Mansouri, K.; Janowska, K.; Stalls, V.; Gobeil, S. M. C.; Kopp, M.; Li, D.; Parks, R.; Hsu, A. L.; Borgnia, M. J.; Haynes, B. F.; Acharya, P.
Cryo-EM analysis of the post-fusion structure of the SARS-CoV spike glycoprotein
Fan, X.; Cao, D.; Kong, L.; Zhang, X.
Potently neutralizing and protective human antibodies against SARS-CoV-2
Zost, S. J.; Gilchuk, P.; Case, J. B.; Binshtein, E.; Chen, R. E.; Nkolola, J. P.; Schäfer, A.; Reidy, J. X.; Trivette, A.; Nargi, R. S.; Sutton, R. E.; Suryadevara, N.; Martinez, D. R.; Williamson, L. E.; Chen, E. C.; Jones, T.; Day, S.; Myers, L.; Hassan, A. O.; Kafai, N. M.; Winkler, E. S.; Fox, J. M.; Shrihari, S.; Mueller, B. K.; Meiler, J.; Chandrashekar, A.; Mercado, N. B.; Steinhardt, J. J.; Ren, K.; Loo, Y. -M.; Kallewaard, N. L.; McCune, B. T.; Keeler, S. P.; Holtzman, M. J.; Barouch, D. H.; Gralinski, L. E.; Baric, R. S.; Thackray, L. B.; Diamond, M. S.; Carnahan, R. H.; Crowe Jr, J. E.
Cao, Y.; Su, B.; Guo, X.; Sun, W.; Deng, Y.; Bao, L.; Zhu, Q.; Zhang, X.; Zheng, Y.; Geng, C.; Chai, X.; He, R.; Li, X.; Lv, Q.; Zhu, H.; Deng, W.; Xu, Y.; Wang, Y.; Qiao, L.; Tan, Y.; Song, L.; Wang, G.; Du, X.; Gao, N.; Liu, J.; Xiao, J.; Su, X. -D.; Du, Z.; Feng, Y.; Qin, C.; Qin, C.; Jin, R.; Xie, X. S.
Structural basis for inhibition of the RNA-dependent RNA polymerase from SARS-CoV-2 by remdesivir
Yin, W.; Mao, C.; Luan, X.; Shen, D. -D.; Shen, Q.; Su, H.; Wang, X.; Zhou, F.; Zhao, W.; Gao, M.; Chang, S.; Xie, Y. -C.; Tian, G.; Jiang, H. -W.; Tao, S. -C.; Shen, J.; Jiang, Y.; Jiang, H.; X.u, Y.; Zhang, S.; Xu, H. E.
Langer, L. M.; Gat, Y.; Bonneau, F.; Conti, E.
Structure of replicating SARS-CoV-2 polymerase
Hillen, H. S.; Kokic, G.; Farnung, L.; Dienemann, C.; Tegunov, D.; Cramer, P.
Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein
Walls, A. C.; Park, Y. -J.; Tortorici, M. A.; Wall, A.; McGuire, A. T.; Veesler, D.
Structural basis for the recognition of the SARS-CoV-2 by full-length human ACE2
Yan, R.; Zhang, Y.; Li, Y.; Xia, L.; Guo, Y.; Zhou, Q.
Structure, function and antigenicity of the SARS-CoV-2 spike glycoprotein
Walls, A. C.; Park, Y. -J.; Tortorici, M. A.; Wall, A.; McGuire, A. T.; Veesler, D
Celia, H.; Botos, I.; Ni, X.; Fox, T.; De Val, N.; Lloubes, R.; Jiang, J.; Buchanan, S. K.
Improved applicability and robustness of fast cryo-electron tomography data acquisition
Eisenstein, F.; Danev, R.; Pilhofer, M.
Solving a new R2lox protein structure by microcrystal electron diffraction
Xu, H.; Lebrette, H.; Clabbers, M. T. B.; Zhao, J.; Griese, J. J.; Zou, X.; Hogbom, M.
Surpassing the physical Nyquist limit to produce super-resolution cryo-EM reconstructions
Feathers, J. R.; Spoth, K. A.; Fromme, J. C.
Single particle cryo-EM reconstruction of 52 kDa streptavidin at 3.2 Angstrom resolution
Fan, X.; Wang, J.; Zhang, X.; Yang, Z.; Zhang, J. -C.; Zhao, L.; Peng, H. -L.; Lei, J.; Wang, H. -W.
Fusion-dependent formation of lipid nanoparticles containing macromolecular payloads
Kulkarni, J. A.; Witzigmann, D.; Leung, J.; van der Meel, R.; Zaifman, J.; Darjuan, M. M.; Grisch-Chan, H. M.; Thöny, B.; Tam, Y. Y. C.; Cullis, P. R.
Putti, M.; Stassen, O. M. J. A.; Schotman, M. J. G.; Sahlgren, C. M.; Dankers, P. Y. W.
A 3.8 Å resolution cryo-EM structure of a small protein bound to an imaging scaffold
Liu, Y.; Huynh, D. T.; Yeates, T. O.
In situ structures of polar and lateral flagella revealed by cryo-electron tomography
Zhu, S.; Schniederberend, M.; Zhitnitsky, D.; Jain, R.; Galán, J. E.; Kazmierczak, B. I.; Liu, J.
Refinement and analysis of the mature Zika virus cryo-EM structure at 3.1 Å resolution
Sevvana, M.; Long, F.; Miller, A. S.; Klose, T.; Buda, G.; Sun, L.; Kuhn, R. J.; Rossmann, M. G.
Xu, H. Lebrette, H.; Yang, T.; Hovmoller, S.; Hogbom, M.; Zou, X.
Cryo-EM structure of 5-HT3A receptor in its resting conformation
Basak, S.; Gicheru, Y.; Samanta, A.; Molugu, S. K.; Huang, W.; la de Fuente, M.; Hughes, T.; Taylor, D. J.; Nieman, M. T.; Moiseenkova-Bell, V.; Chakrapani, S.
関連資料
Nyquist frequency
Dose fractionation and motion correction
Improving DQE with counting and super-resolution
| Modulation transfer function (MTF) curves | ||
|---|---|---|
| 200 kV | 300 kV | |
|
K3 |
CDS | CDS |
| Standard | Standard | |
| K2 | Standard | Standard |