K3® カメラの性能は直接検出型カメラの撮像能力の新たなベンチマークとなります。全てを見直し新たに設計することで、ライフサイエンスとマテリアルサイエンス双方の分野に求められる低電子線照射量観察に最適化されたカメラです。リアルタイム、単電子カウンティングを実現するために、Gatanの全ての知見を投入して開発された直接検出型カメラがK3カメラです。
- 強力なインラインの信号処理系によって K2® を超えるDQEを実現。CDSモードの追加によりDQEはさらに向上
- リアルタイムの電子カウンティング処理によって試料の良否を即座に判断
- オプションのインライン、GPUベースのモーションコレクション機能によってテラバイト級の生データが不要
- 1500フレーム/秒 - K2カメラ比3.75倍
- アプリケーションに適した視野範囲が選択可能
- 24 メガピクセル – 最高性能の電子顕微鏡のスループットを最大化
- 14 メガピクセル – スクリーニング用電子顕微鏡をデータ取得用の電子顕微鏡へと機能強化
-
 K3 camera for MicroED/3DED
K3 camera for MicroED/3DED
-
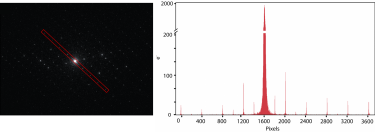 Electron-counted SAED of ZSM-5 with the K3® camera
Electron-counted SAED of ZSM-5 with the K3® camera
-
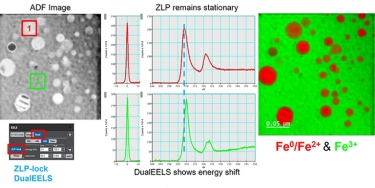
 DualEELS: The importance of low-loss correction of electron energy-loss spectroscopy data
DualEELS: The importance of low-loss correction of electron energy-loss spectroscopy data
-
 Imaging molecules in their native environment: Cryo-electron tomography of PCDH15 complexes in mouse stereocilia
Imaging molecules in their native environment: Cryo-electron tomography of PCDH15 complexes in mouse stereocilia
-
 Cryo EM reveals mechanisms of gating and drug modulation in 5 HT3A receptors webinar
Cryo EM reveals mechanisms of gating and drug modulation in 5 HT3A receptors webinar
-
 CryoARM / K3の組み合わせによる高分解能観察 : SerialEMとLatitude、そしてデータ取得の将来
CryoARM / K3の組み合わせによる高分解能観察 : SerialEMとLatitude、そしてデータ取得の将来
-

 構造生物学を用いたパンデミックへの備え
構造生物学を用いたパンデミックへの備え
-

 NUANCE Workshop on 4D STEM: Fundamentals of Electron Diffraction and 4D STEM
NUANCE Workshop on 4D STEM: Fundamentals of Electron Diffraction and 4D STEM
-

 NUANCE Workshop on 4D STEM: Data Processing in DM
NUANCE Workshop on 4D STEM: Data Processing in DM
-

 NUANCE Workshop on 4D STEM: Data Processing using Python
NUANCE Workshop on 4D STEM: Data Processing using Python
In situ structural analysis of SARS-CoV-2 spike reveals flexibility mediated by three hinges
Turoňová, B.; Sikora, M.; Schürmann, C.; Hagen, W. J. H.; Welsch, S.; Blanc, F. E. C.; von Bülow, S.; Gecht, M.; Bagola, K.; Hörner, C.; van Zandbergen, G.; Landry, J.; de Azevedo, N. T. D.; Mosalaganti, S.; Schwarz, A.; Covino, R.; Mühlebach, M. D.; Hummer, G.; Locker, J. K.; Beck, M.
Molecular architecture of the SARS-CoV-2 virus
Yao, H.; Song, Y.; Chen, Y.; Wu, N.; Xu, Sun, C.; Zhang, J.; Weng, T.; Zhang, Z.; Wu, Z.; Cheng, L,; Shi, D.; Lu, X.; Lei, J.; Crispin, M.; Shi, Y.; Li, L.; Li, S.
Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation
Wrapp, D.; Wang, N.; Corbett, K. S.; Goldsmith, J. A.; Hsieh, C. -L.; Abiona, O.; Graham, B. S.; McLellan, J. S.
Electron-counting MicroED data with the K2 and K3 direct electron detectors
Clabbers, M. T. B.; Martynawycz, M. W.; Hattne, J.; Nannenga, B. L.; Gonen, T.
Time-resolved cryo-EM using a combination of droplet microfluidics with on-demand jetting
Torino, S.; Dhurandhar, M.; Stroobants, A.; Claessens, R.; Efremov, R. G.
Szemán, A. J. K.; Stráner, P.; Jákli, I.; Hosogi, N.; Harmat, V.; Menyhárd, D. K.; Perczel, A.
Structural basis of actin filament assembly and aging
Oosterheert, W.; Klink, B. U.; Belyy, A.; Pospich, S.; Raunser, S.
Helical ultrastructure of the metalloprotease meprin α in complex with a small molecule inhibitor
Bayly-Jones, C.; Lupton, C. J.; Fritz, C.; Venugopal, H.; Ramsbeck, D.; Wermann, M.; Jäger,C.; Marco, A. D.; Schilling, S.; Schlenzig, D.; Whisstock, J. C.
Stern, A. M.; Yang, Y.; Meunier, A. L.; Liu, W.; Cai, Y.; Ericsson, M.; Liu, L.; Goedert, M.; Scheres, S. H. W.; Selkoe, D.J
Cryo-EM structure of the human NKCC1 transporter reveals mechanisms of ion coupling and specificity
Neumann, C.; Rosenbæk, L. L.; Flygaard, R. K.; Habeck, M.; Karlsen, J. L.; Wang, Y.; Larsen, K. L.; Gad, H. H.; Hartmann, R.; Lyons, J. A; Fenton, R. A.; Nissen, P.
Algal photosystem I dimer and high-resolution model of PSI-plastocyanin complex
Naschberger, A.; Mosebach, L.; Tobiasson, V.; Kuhlgert, S.; Scholz, M.; Perez-Boerema, A.; Ho, T. T. H.; Vidal-Meireles, A.; Takahashi, Y.; Hippler, M.; Amunts, A.
Chang, M. R.; Tomasovic, L.; Kuzmina, N. A.; Ronk, A. J.; Byrne, P. O.; Johnson, R.; Storm, N.; Olmedillas, E.; Hou, Y. J.; Schäfer, A.; Leist, S. R.; Tse, L. T.; Ke, H.; Coherd, C.; Nguyen, K.; Kamkaew, M.; Honko, A.; Zhu, Q.; Alter, G.; Saphire, E. O.; McLellan, J. S.; Griffiths, A.; Baric, R. S.; Bukreyev. A.; Marasco. W. A.
Frieg, B.; Geraets, J. A.; Strohäker, T.; Dienemann, C.; Mavroeidi, P.; Jung, B. C.; Kim, W. S.; Lee, S. J.; Xilouri, M.; Zweckstetter. M.; Schröder. G. F.
Lyu, M.; Ayala, J. C.; Chirakos, I.; Su, C. -C.; Shafer, W. M.; Yu, E. W.
Organic crystal growth: Hierarchical self-assembly involving nonclassical and classical steps
Biran, I.; Rosenne, S.; Weissman, H.; Tsarfati, Y.; Houben, L.; Rybtchinski. B.
Structure of the PAPP-ABP5 complex reveals mechanism of substrate recognition
Judge, R. A.; Sridar, J.; Tunyasunvunakool, K.; Jain, R.; Wang, J. C. K.; Ouch, C.; Xu, J.; Mafi, A.; Nile, A. H.; Remarcik, C.; Smith, C. L.; Ghosh, C.; Xu, C.; Stoll, V.; Jumper, J.; Singh, A. H.; Eaton, D.; Hao, Q.
Deagglomeration of DNA nanomedicine carriers using controlled ultrasonication
Hinchliffe, B. A.; Turner, P.; J. H. Cant, D.; De Santis, E.; Aggarwal, P.; Harris, R.; Templeton, D.; Shard, A. G.; Hodnett, M.; Minelli, C.
Structures of a phycobilisome in light-harvesting and photoprotected states
Domínguez-Martín, M. A.; Sauer, P. V.; Kirst, H.; Sutter, M.; Bína, D.; Greber, B. J.; Nogales, E. ;Polívka, T.; Kerfeld, C. A.
Zhu, J.; Huang, W.; Zhao, J., Huynh, L.; Taylor, D. J.; Harris, M. E.
Miyakawa, T.; Yang, J.; Kawasaki, M.; Adachi, N.; Fujii, A.; Miyauchi, Y.; Muramatsu, T.; Moriya, T.; Senda, T.; Tanokura, M.
Zhao, J.; Makhija, S.; Zhou, C.; Zhang, H.; Wang, Y.; Muralidharan, M.; Huang, B.; Cheng, Y.
Mechanistic details of CRISPR-associated transposon recruitment and integration revealed by cryo-EM
Park, J. U.; Tsai, A. W. -T.; Chen, T. H.; Peters, J. E.; Kellogg, E. H.
Specific recognition and ubiquitination of slow-moving ribosomes by human CCR4-NOT
Absmeier, E.; Chandrasekaran, V.; O'Reilly, F. J.; Stowell, J. A. W.; Rappsilber, J.; Passmore, L. A.
Cryo-electron microscopy of extracellular vesicles
Cai, K.; Sibert, B. S.; Kumar, A.; Yang, J.; Larson, M.; Thompson, K.; Wright, E. R.
Peck, J. V.; Strauss, J. D.; Fay, J. F.
Cryo-EM structure of an active bacterial TIR–STING filament complex
Morehouse, B. R.; Yip, M. C. J.; Keszei, A. F. A.; McNamara-Bordewick, N. K.; Shao, S.; Kranzusch, P. J.
Maintaining the momentum in cryoEM for biological discovery
Thompson, R.; Halfon, Y.; Aspinall, L.; White, J.; Hirst, I. J.; Wang, Y.; Darrow, M.; Muench, S. P.
Structural analysis of the basal state of the Artemis:DNA-PKcs complex
Watanabe, G.; Lieber, M. R.; Williams, D. R.
Flipped over U: structural basis for dsRNA cleavage by the SARS-CoV-2 endoribonuclease
Frazier, M. N.; Wilson, I. M.; Krahn, J. M.; Butay, K. J.; Dillard, L. B.; Borgnia, M. J.; Stanley, R. E.
Electron-counting MicroED data with the K2 and K3 direct electron detectors
Clabbers, M. T. B.; Martynowycz, M. W.; Hattne, J.; Nannenga, B. L.; Gonen, T.
Structures and gating mechanisms of human bestrophin anion channels
Owji, A. P.; Wang, J.; Kittredge, A.; Clark, Z.; Zhang, Y.; Hendrickson, W. A.; Yang, T.
Structural insights into dsRNA processing by Drosophila Dicer-2–Loqs-PD
Su, S.; Wang, J.; Deng, T.; Yuan, X.; He, J.; Liu, N.; Li, X.; Huang, Y.; Wang, H. -W.; Ma, J.
Structures and mechanism of the plant PIN-FORMED auxin transporter
Ung, K. L.; Winkler, M.; Schulz, L.; Kolb, M.; Janacek, D. P.; Dedic, E.; Stokes, D. L.; Hammes, U. Z.; Pedersen, B. P.
Cryo-EM structures of SARS-CoV-2 Omicron BA.2 spike
Stalls, V.; Lindenberger, J.; Gobeil, S. M. -C.; Henderson, R.; Parks, R.; Barr, M.; Deyton, M.; Martin, M.; Janowska, K.; Huang, X.; May, A.; Speakman, M.; Beaudoin, E.; Kraft, B.; Lu, X.; Edwards, R. J.; Eaton, A.; Montefiori, D. C.; Williams, W. B.; Saunders, K. O.; Wiehe, K.; Haynes, B. F.; Acharya, P.
Structure and flexibility of the yeast NuA4 histone acetyltransferase complex
Zukin, S. A.; Marunde, M. R.; Popova, I. K.; Nogales, E.; Patel, A. B.
Structure and flexibility of the yeast NuA4 histone acetyltransferase complex
Zukin, S. A.; Marunde, M. R.; Popova, I. K.; Nogales, E.; Patel, A. B.
Role of aIF5B in archaeal translation initiation
Kazan, R.; Bourgeois, G.; Lazennec-Schurdevin, C.; Larquet, E.; Mechulam, Y.; Coureux, P. -D.; Schmitt, E.
Ion complexation waves emerge at the curved interfaces of layered minerals
Whittaker, M. L.; Ren, D.; Ophus, C.; Zhang, Y.; Waller, L.; Gilbert, B.; Banfield, J. F.
Compact IF2 allows initiator tRNA accommodation into the P site and gates the ribosome to elongation
Basu, R. S.; Sherman, M. B.; Gagnon, M. G.
Structural Basis for pH-gating of the K+ channel TWIK1 at the selectivity filter
Turney, T. S.; Li, V.; Brohawn, S. G.
Cryo-EM structures of SARS-CoV-2 Omicron BA.2 spike
Stalls, V.; Lindenberger, J.; Gobeil, S. M. -C.; Henderson, R.; Parks, R.; Barr, M.; Deyton, M.; Martin, M.; Janowska, K.; Huang, X.; May, A.;l Speakman, M.; Beaudoin, E.; Kraft, B.; Lu, X.; Edwards, R. J.; Eaton, A.; Montefiori, D. C.; Williams, W.; Saunders, K. O.; Wiehe, K.; Haynes, B. F.; Acharya, P.
Structure of S1PR2–heterotrimeric G13 signaling complex
Chen, H.; Chen, K.; Huang, W.; Staudt, L. M.; Cyster, J. G.; Li, X.
Ishii, N.
Structure of the type V-C CRISPR-Cas effector enzyme
Kurihara, N.; Nakagawa, R.; Hirano, H.; Okazaki, S.; Tomita, A.; Kobayashi, K.; Kusakizako, T.; Nishizawa, T.; Yamashita, K.; Scott, D. A.; Nishimasu, H.; Nureki, O.
Zhao, W.; Jensen, G. J.
The giant Mimivirus 1.2 Mb genome is elegantly organized into a 30 nm helical protein shield
Villalta, A.; Schmitt, A.; Estrozi, L. F.; Quemin, E. R. J.; Alempic, J. -M.; Lartigue, A.; Pražák, V.; Belmudes, L.; Vasishtan, D.; Colmant, A. M. G.; Honoré, F. A.; Couté, Y.; Grünewald, K.; Abergel, C.
Structural and functional impact by SARS-CoV-2 Omicron spike mutations
Zhang, J.; Cai, Y.; Lavine, C. L.; Peng, H.; Zhu, H.; Anand, K.; Tong, P.; Gautam, A.; Mayer, M. L.; Rits-Volloch, S.; Wang, S.; Sliz, P.; Wesemann, D. R.; Yang, W.; Seaman, M. S.; Lu, J.; Xiao, T.; Chen, B.
Yu, H.; Hamaguchi, T.; Nakajima, Y.; Kato, K.; Kawakami, K.; Akita, F.; Yonekura, K.; Shen, J. -R.
Structure-guided multivalent nanobodies block SARS-CoV-2 infection and suppress mutational escape
Koenig, P. -D.; Das, H.; Liu H.; Kümmerer, B. M.; Gohr, F. N.; Jenster, L. -M.; Schiffelers, L. D. J.; Tesfamariam, Y. M.; Uchima, M.; Wuerth, J. D.; Gatterdam, K.; Ruetalo, N.; Christensen, M. H.; Fandrey, C. I.; Normann, S.; Tödtmann, J.; M. P.; Pritzl, S.; Hanke, L.; Boos, J.; Yuan, M.; Zhu, X.; Schmid-Burgk, J. L.; Kato, H.; Schindler, M.; Wilson, I. A.; Geyer, M.; Ludwig, K. U.; Hällberg, M.; Wu, N. C.; Schmidt, F. I.
Ultrapotent antibodies against diverse and highly transmissible SARS-CoV-2 variants
Wang, L.; Zhou, T.; Zhang, Y.; Yang, E. S.; Schramm, C. A.
Native-like SARS-CoV-2 spike glycoprotein expressed by ChAdOx1 nCoV-19/AZD1222 vaccine
Watanabe, Y.; Mendonça, L.; Allen, E. R.; Howe, A.; Lee, M.; Allen, J. D.; Chawla, H.; Pulido, D.; Donnellan, F.; Davies, H.; Ulaszewska, M.; Belij-Rammerstorfer, S.; Morris, S.; Krebs, A. -S.; Dejnirattisai, W.; Mongkolsapaya, J.; Supasa, P.; Screaton, G. R.; Green, C. M.; Lambe, T.; Zhang, P.; Gilbert, S. C.; Crispin, M.
Structure of a microtubule-bound axonemal dynein
Walton, T.; Wu, H.; Brown, A.
Mechanism of SARS-CoV-2 polymerase stalling by remdesivir
Kokic, G.; Hillen, H. S.; Tegunov, D.; Dienemann, C.; Seitz, F.; Schmitzova, J.; Farnung, L.; Siewert, A.; Höbartner, C.; Cramer, P.
Stabilizing the closed SARS-CoV-2 spike trimer
Juraszek, J.; Rutten, L.; Blokland, S.; Bouchier, P.; Voorzaat, R.; Ritschel, T.; Bakkers, M. J. G.; Renault , L. L. R.; Langedijk, J. P. M.
Zhou, T.; Tsybovsky, Y.; Gorman, J.; Rapp, M.; Cerutti, G.; Chuang, G. -Y.; Katsamba, P. S.; Sampson, J. M.; Schön, A.; Bimela, J.; Boyington, J. C.; Nazzari, A.; Olia, A. S.; Shi, W.; Sastry, M.; Stephens, T.; Stuckey, J.; Teng, I. -T.; Kwong, P. D
Architecture of a SARS-CoV-2 mini replication and transcription complex
Yan, L.; Zhang, Y.; Ge, J.; Zheng, L.; Gao, Y.; Wang, T.; Jia, Z.; Wang, H.; Huang, Y.; Li, M.; Wang, Q.; Ra, Z.; Lou, Z.
An ultrapotent synthetic nanobody neutralizes SARS-CoV-2 by stabilizing inactive spike
Schoof, M.; Faust, B.; Saunders, R. A.; Sangwan, S.; Rezelj, V.; Hoppe, N.; Boone, M.; Billesbølle, C. B.; Puchades, C.; Azumaya, C. M.; Kratochvil, H. T.; Zimanyi, M.; Deshpande, I.; Liang, J.; Dickinson, S.; Nguyen, H. C.; Chio, C. M.; Merz, G. E.; Thompson, M. C.; Diwanji, D.; Schaefer, K.; Anand, A. A.; Dobzinski, N.; Zha, B. S.; Simoneau, C. R.; Leon, K.; White, K. M.; Chio, U. S.; Gupta, M.; Jin, M.; Li, F.; Liu, Y.; Zhang, K.; Bulkley, D.; Sun, M.; Smith, A. M.; Rizo, A. N.; Moss, F.; Brilot, A. F.; Pourmal, S.; Trenker, R.; Pospiech, T.; Gupta, S.; Barsi-Rhyne, B.; Belyy, V.; Barile-Hill, A. W.; Nock, S.; Liu, Y.; Krogan, N. J.; Ralston, C. Y.; Swaney, D. L.; García-Sastre, A.; Ott, M.; Vignuzzi, M.; QCRG Structural Biology Consortium; Walter, P.; Manglik, A.
Zhou, T.; Teng, I. -T.; Olia, A. S.; Cerutti, G.; Gorman, J.; Nazzari, A.; Shi, W.; Tsybovsky, Y.; Wang, L.; Wang, S.; Zhang, B.; Zhang, Y.; Katsamba, P. S.; Petrova, Y.; Banach, B. B.; Fahad, A. S.; Liu, L.; Lopez Acevedo, S. N.; Madan, B.; de Souza, M. O.; Pan, X.; Wang, P.; Wolfe, J. R.; Yin, M.; Ho, D. D.; Phung, E.; DiPiazza, A.; Chang, L. A.; Abiona, O. M.; Corbett, K. S.; DeKosky, B. J.; Graham, B. S.; Mascola, J. R.; Misasi, J.; Ruckwardt, T.; Sullivan, N. J.; Shapiro, L.; Kwong, P. D.
Langer, L. M.; Gat, Y.; Bonneau, F.; Conti, E.
Structural basis for the recognition of the SARS-CoV-2 by full-length human ACE2
Yan, R.; Zhang, Y.; Li, Y.; Xia, L.; Guo, Y.; Zhou, Q.
Improved applicability and robustness of fast cryo-electron tomography data acquisition
Eisenstein, F.; Danev, R.; Pilhofer, M.
Surpassing the physical Nyquist limit to produce super-resolution cryo-EM reconstructions
Feathers, J. R.; Spoth, K. A.; Fromme, J. C.
Applications
モデル1025, 1024
データシート
関連製品
BioContinuum®イメージングフィルタ
Alpine™ 直接検出カメラ
K3 IS カメラ
Latitude® S ソフトウェア
Elsa™クライオトランスファーホルダー
関連資料
ナイキスト周波数
線量分割および視野移動補正
カウンティングと超解像を用いたDQEの向上
| Modulation transfer function (MTF) curves | ||
|---|---|---|
| 200 kV | 300 kV | |
|
K3 |
CDS | CDS |
| Standard | Standard | |
ACKNOWLEDGMENT
Continuing our prosperous collaborations that built the K2, the K3 is the successful result of Peter Denes' team at Lawrence Berkeley National Laboratory and David Agard.

